Le monochromatisme à cônes bleus (BCM) est une maladie rétinienne génétique rare, dont l’incidence est estimée à 1/100.000. Il s’agit d’une dégénérescence rétinienne héréditaire (DRH) due à des mutations du cluster de gènes OPN1LW/OPN1MW, codant pour des opsines sensibles aux longueurs d’ondes longues et moyennes (L et M, respectivement de couleur rouge et verte) des cellules photoréceptrices des cônes dans la rétine. Les handicaps visuels sont graves et s’expriment par une acuité visuelle réduite, une perception anormale des couleurs, une myopie, un nystagmus et une photophobie [11,20,21,34,52].
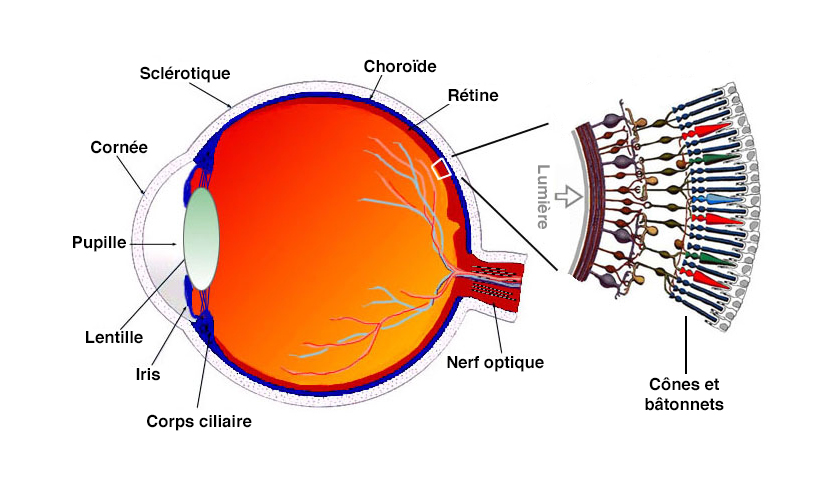
Le monochromatisme à cônes bleus affecte la rétine, à l’arrière de l’œil.
Du fait qu’il s’agit d’une pathologie liée à l’X récessif, elle frappe presque exclusivement les hommes (XY), tandis que les femmes porteuses (XX) ne montrent que rarement certains symptômes, d’ailleurs atténués.
Le monochromatisme à cônes bleus est généralement considéré comme une maladie stationnaire, dont les symptômes se manifestent d’abord dès la prime enfance ; néanmoins, l’on observe chez de nombreux patients une progression de la maladie, avec des changements maculaires associés à une dégénération maculaire [5,6,34,52]. Le premier symptôme observé est le nystagmus chez des bébés de 2 à 3 mois.
Vidéo d’un bébé de 4 mois atteint de monochromatisme à cônes bleus et présentant un nystagmus.
Symptômes
La rétine humaine possède trois types de cônes, qui assurent la vision diurne, l’acuité visuelle et la vision des couleurs : ils sont sensibles à la lumière à ondes longues (cônes rouges), moyennes (cônes verts) et courtes (cônes bleus) [41]. Lorsqu’une personne souffre de monochromatisme à cônes bleus, il y a un dysfonctionnement des cônes rouges et verts, tandis que les cônes bleus fonctionnent normalement [5,38,39]. Les signes et symptômes peuvent inclure une faible acuité visuelle (en termes de clarté ou de netteté), une compromission de la vision des couleurs, une photophobie (sensibilité à la lumière), une myopie et un nystagmus (mouvements rapides et involontaires de l’œil).
Différents symptômes caractérisent le monochromatisme à cônes bleus :
- Acuité visuelle réduite, comprise entre 1/10ème et 3/10èmes.
- Faible différenciation des couleurs (incapacité partielle ou totale à distinguer entre les différentes couleurs)
- Photophobie (intolérance à la lumière), associée à un trouble de la vision en pleine lumière, spécialement pendant la journée.
- Myopie – les patients sont souvent myopes
- Nystagmus (caractérisé par des mouvements rythmiques et incontrôlés de l’œil), qui se présente dès l’âge de 2 mois et peut s’atténuer graduellement avec l’âge.
Pour la plupart des individus atteints de monochromatisme à cônes bleus, les symptômes sont généralement stationnaires. Néanmoins, certaines études cliniques font état d’une progression de la maladie, avec des changements maculaires [6,34,52].

Un enfant malvoyant, contraint de se pencher de très près sur son livre
Synonymes :
- Monochromatisme à cônes bleus ;
- Monochromatisme à cônes S ;
- Achromatopsie atypique liée à l’X ;
- Achromatopsie incomplète liée à l’X.
Causes : gènes et mutations
Trois gènes sont impliqués dans le monochromatisme à cônes bleus ; ils sont situés au point Xq28, à l’extrémité du bras q du chromosome X. Ces trois gènes sont en tandem et sont :
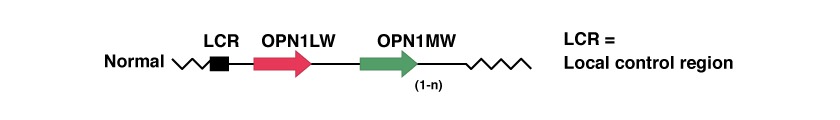
Ces gènes codent pour des protéines qui sont nécessaires pour convertir la lumière en signaux électriques que le cerveau utilise pour traiter les informations visuelles. Ces protéines sont appelées opsines des cônes L et M et jouent un rôle fondamental dans ce traitement.
La région de contrôle du locus (LCR) agit comme promoteur de l’expression des premiers des deux gènes suivants, OPN1LW et OPN1MW, en codant pour les opsines responsables de la capture de la lumière (respectivement rouge et verte) dans la rétine. En outre, la LCR assure qu’un seul des deux gènes opsine (rouge ou vert) soit exprimé de manière exclusive dans chaque cône [49,54,55].
Il existe de nombreuses mutations génétiques pouvant intéresser ce groupe de gènes et mener ainsi au BCM [9,17,25,26,28,38,39] : une délétion de la LCR, la délétion intragénique des exons dans les gènes OPN1LW et OPN1MW et un mécanisme en deux étapes avec recombinaison homologue et inactivation ponctuelle.
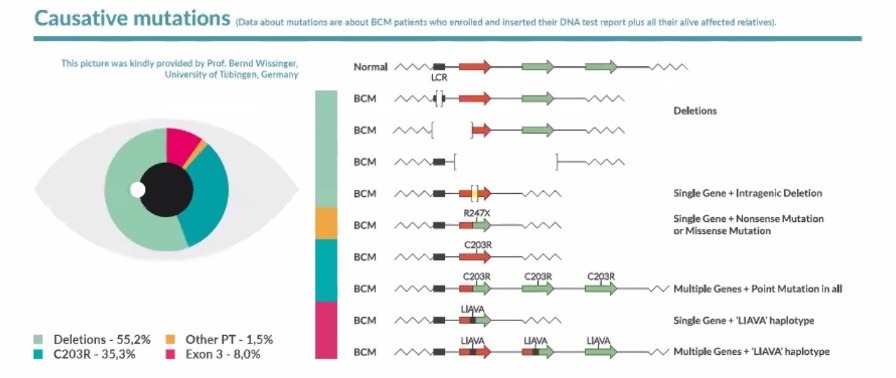
Image tirée des données agrégées du Registre des patients (Patient Registry) – BCMRegistry-Poster-2022-Final – [1]. Le BCM Patient Registry est disponible sur www.BCMRegistry.org

Nous pouvons vous aider à mieux comprendre.
N’hésitez pas à nous écrire ou à rejoindre notre communauté en ligne.
Comment le BCM se transmet-il ?
Le monochromatisme à cônes bleus se transmet génétiquement, par le biais de gènes qui passent des parents aux enfants.
Plus précisément, le monochromatisme à cônes bleus est une maladie liée à l’X récessif, ce qui signifie que la maladie s’exprime chez les hommes (XY) qui sont hémizygotes pour la mutation génétique et chez les rares femmes qui sont homozygotes pour la mutation génétique, c’est-à-dire chez les femmes qui présentent sur les deux chromosomes X la mutation génétique en question. Il n’est donc pas impossible pour une femme (XX) d’être atteinte de monochromatisme à cônes bleus, mais c’est extrêmement rare.
Diagnostic
Chez un enfant de sexe masculin, la présence, dès l’âge de 2 mois, d’une aversion à la lumière et d’un nystagmus peut laisser supposer un cas de monochromatisme à cônes bleus, mais ne fournit pas assez d’indications pour qu’il soit possible d’en établir la forme. Pour identifier un cas de BCM, il est nécessaire de reconstituer l’histoire familiale, avec, si le cas n’est pas unique dans la famille, la condition liée à la transmission du chromosome X. Chez les adultes, il est possible de tester l’acuité visuelle et la vision des couleurs et d’établir un diagnostic clinique. Pour autant, la phase la plus importante consiste à obtenir une confirmation génétique par le biais d’un test ADN.
Les instruments diagnostiques les plus indiqués sont :
- Un test ADN.
- Un test des couleurs (par exemple, Farnsworth D-15 ou Farnsworth Munsell 100 Hue).
- La reconstitution de l’histoire familiale ou du pedigree familial de la maladie.
- L’électrorétinogramme (ERG), qui peut démontrer la perte des fonctions des cônes L/M avec le maintien subséquent de la fonction des cônes S et des bâtonnets [4].
Une démarche importante consiste à effectuer un diagnostic différentiel, en faisant la distinction entre le monochromatisme à cônes bleus et d’autres maladies présentant des caractéristiques cliniques similaires, comme par exemple l’achromatopsie [7]. Il est fondamental d’établir le bon diagnostic, car l’évolution de la maladie, les traitements possibles et les dispositifs d’aide nécessaires changent d’une pathologie à l’autre. L’étape cruciale pour confirmer le diagnostic du monochromatisme à cônes bleus est d’effectuer le test ADN.
Thérapie génique et Traitements
À l’heure actuelle, il n’existe pas de traitement connu pour le monochromatisme à cônes bleus ; quoi qu’il en soit, l’efficacité et la sécurité de traitements potentiels sont en cours d’évaluation, dont le plus prometteur est la thérapie génique.
L’objectif que se fixent les études sur la thérapie génique [15,16,23,51,59,61,62] est d’introduire, par voie virale, dans les cellules rétiniennes exprimant des gènes mutants associés avec le phénotype du monochromatisme à cônes bleus, des formes saines du gène OPN1LW, permettant ainsi aux cellules photoréceptrices rétiniennes de se réparer et de fonctionner correctement, en réponse aux instructions associées au gène sain ajouté.
Par ailleurs, des aides visuelles correctives et une thérapie visuelle personnalisée fournies par des spécialistes de la malvoyance peuvent aider les patients à corriger l’effet d’éblouissement et à optimiser leur acuité visuelle.
Épidémiologie
Le monochromatisme à cônes bleus est une cause de malvoyance héréditaire dont l’incidence est estimée à environ 1/100.000 personnes [27]. Cette maladie frappe les individus de sexe masculin intéressés par la mutation liée à l’X, tandis que les individus de sexe féminin restent généralement des porteuses saines de BCM.
Histoire
Les premières découvertes
Le monochromatisme à cônes bleus est une pathologie connue depuis de nombreuses années, la première description détaillée remontant à 1777, lorsque Huddart [24] observa que les individus atteints de monochromatisme à cônes bleus avaient des difficultés à distinguer les couleurs, mais pouvaient faire la différence entre le blanc, le noir et différentes couleurs claires ou vives.
Par la suite, des études sur le BCM furent menées par Sloan en 1954 [48] et Blackwell et Blackwell en 1961 [8], qui décrivirent des patients capables de distinguer entre des signaux bleus et jaunes, qui semblaient avoir des bâtonnets et des cellules de cônes S fonctionnels. En outre, Spivey, en 1965 [50], indiqua que les individus atteints étaient en mesure de voir de petits objets bleus sur un grand fond jaune et inversement.
La maladie a également été étudiée par Alpern et al. (1960) [2,3] et par Fleischman (1981), mais les résultats les plus importants ont été obtenus en 1989 et 1993 par Nathans et al. [38,39] et en 1991 par Reyniers et al. [46], qui ont identifié les gènes responsables du monochromatisme à cônes bleus.
Études récentes
Ce n’est que ces dernières années, et grâce au soutien de la BCM Families Foundation (BCMFF), que les études portant sur le monochromatisme à cônes bleus se sont orientées vers la recherche d’un traitement.
En particulier, la BCMFF finance depuis 2010 des études cliniques à l’Université de Pennsylvanie, en vue de comprendre si la thérapie génique peut aider à soigner le monochromatisme à cônes bleus. Des résultats positifs sans précédents montre une présence suffisante de cellules de cônes dans la rétine pour justifier la thérapie génique [11]. Ces cellules photoréceptrices peuvent en effet être traitées au moyen de la thérapie génique. Une autre étude a comparé des patients relevant des deux principales mutations causant le monochromatisme à cônes bleus (les délétions et les mutations faux-sens C203R) et identifié des fenêtres d’âge pour déterminer l’opportunité d’une intervention par le biais de la thérapie génique [52]. Cette série de recherches constitue une étude d’histoire naturelle du monochromatisme à cônes bleus. D’autres études ont mené à l’identification de résultats de mesure pour une expérimentation clinique et à l’identification de critères d’inclusion et d’exclusion [12,30,33,47,51].
Le soutien financier de la BCM Families Foundation a permis d’effectuer différentes études sur les vecteurs de la thérapie génique. L’équipe du Dr. W.W. Hauswirth à l’Université de Floride, d’abord, puis l’équipe du Dr. Wen Tao Deng, à l’Université de Virginie Occidentale, ont travaillé sur des modèles animaux du monochromatisme à cônes bleus afin d’identifier le meilleur vecteur d’AAV pour la thérapie génique [15,16,18,23,59,61,62]. Adverum Biotechnologies, Inc. a évalué un nouveau vecteur intravitréen de thérapie génique pour le traitement du BMC [23], ADVM-062, un vecteur optimisé pour l’expression spécifique de l’opsine L humaine. Contrairement aux thérapies existantes impliquant l’injection sous-rétinienne d’un vecteur, ADVM-062 peut être administré par simple injection intravitréenne (IVT), qui présente moins de risques pour la structure rétinienne centrale des patients atteints de BCM.
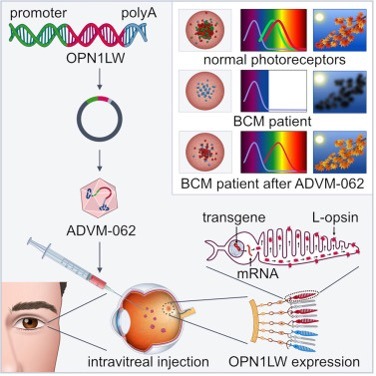
Image tirée de réf. [23]
Plusieurs études scientifiques ont été menées sur le monochromatisme à cônes bleus [9,17,25,26,28, 38,39,46,58] pour identifier toutes les mutations responsables de sa survenue et comprendre en profondeur les principales mutations. Des technologies de séquençage de nouvelle génération (NGS) d’ADN ont été introduites en laboratoire et permettent le séquençage de tout l’exome ou de tout le génome. Ces technologies représentent l’avenir des tests ADN, du fait qu’elles donnent la possibilité de séquencer un exome ou génome en quelques jours, à un coût raisonnable, et permettent de détecter une maladie génétique qui sinon resterait cachée. Toutefois le séquençage de nouvelle génération NGS à courte lecture, largement utilisé, ne se prête pas à l’analyse du cluster génique OPN1LW/OPN1MW du monochromatisme à cônes bleus et un nouvel instrument a récemment été développé [22].
Vidéos
- Vidéo provenant du Low Vision Center (centres de malvoyance) de l’Indiana, montrant des symptômes du monochromatisme à cônes bleus et l’utilisation de lentilles de contact à filtre magenta :
2. Vidéo d’un bébé de 4 mois atteint de monochromatisme à cônes bleus et présentant un nystagmus
Ressources externes:
- The BCM Patient Registry
- OMIM page for Blue Cone Monochromacy – Online Mendelian Inheritance in Man (OMIM) is an online catalog of human genes and genetic disorders developed and maintained by the McKusick-Nathans Institute of Genetic Medicine at Johns Hopkins University.
- Orphanet – Orphanet is a European database and web portal that provides information about rare diseases to promote research and collaboration in the field.
- University of Tübingen, Germany project on Blue Cone Monochromacy
- A Colorful Cure: How Gene Therapy Is Treating Blue Cone Monochromacy (Forbes, 2023)
- Achromatopsia.info – Journey through the Light
- Kohl, S., Hamel, C. Clinical utility gene card for: blue cone monochromatism. Eur J Hum Genet19, 732 (2011)
- National Center for Advancing Translational Sciences
- The University of Arizona Health Sciences’ page for Blue Cone Monochromacy
- Fighting Blindness’ page for Blue Cone Monochromacy
Références
- “Patient Registry – Blue Cone Monochromacy”.
- Alpern M, Lee G B, Maaseidvaag F, Miller SS, (1971). “Colour vision in blue cone ‘monochromacy’”. Physiol. 212 (1): 211–33. doi:10.1113/jphysiol.1971.sp009318.
- Alpern M, Falls H F, Lee G B, (1960). “The enigma of typical total monochromacy”. Am. J. Ophthalmol. 50 (5): 996–1012. doi:1016/0002-9394(60)90353-6. PMID13682677.
- Andréasson, S; Tornqvist, K (1991). “Electroretinograms in patients with achromatopsia”. Acta Ophthal. (Copenh) 69 (6): 711-716. PMID 1789084. https://doi.org/10.1111/j.1755-3768.1991.tb02048.x
- Ayyagari R, Kakuk L E, Bingham E L, Szczesny J J, Kemp J, Toda Y, Felius J, Sieving P A, (2000). “Spectrum of color gene deletions and phenotype in patients with blue cone monochromacy” Genet. 107 (1): 75–82. doi:10.1007/s004390000338.
- Ayyagari R, Kakuk L E, Coats C L, Bingham EL, Toda Y, Felius J, Sieving P A, (1999).”Bilateral macular atrophy in blue cone monochromacy (BCM) with loss of the locus control region (LCR) and part of the red pigment gene”. Mol. Vis. 28: 5-13. PMID 10427103.
- Berson EL, Sandberg MA, Rosner B, Sullivan PL, (1983). “Color plates to help identify patients with blue cone monochromatism”. Am. J. Ophthalmol. 95 (6): 741–7. doi:1016/0002-9394(83)90058-2. PMID6602551.
- Blackwell H R, Blackwell O M, (1961). “Rod and cone receptor mechanisms in typical and atypical congenital achromatopsia”. Vision Res. 1 (1–2): 62–107. doi:1016/0042-6989(61)90022-0.
- Buena-Atienza E, Nasser F, Kohl S, Wissinger B, (2018) “A 73,128 bp de novo deletion encompassing the OPN1LW/OPN1MW gene cluster in sporadic Blue Cone Monochromacy: A case report.” BMC Med. Genet. 19, 107 (2018). – DOI: 1186/s12881-018-0623-8
- Carroll J, et al. (2010) “Deletion of the X-linked opsin gene array locus control region (LCR) results in disruption of the cone mosaic” Vision Res. 50, 1989–1999 (2010). – doi: 1016/j.visres.2010.07.009
- Cideciyan A V, Hufnagel R B, Carroll J, Sumaroka A, Luo X, Schwartz S B, Dubra A, Land M, Michaelides M, Gardner J C, Hardcastle A J, Moore A T, Sisk R A, Ahmed Z M, Kohl S, Wissinger B, Jacobson S G, (2013) “Human cone visual pigment deletions spare sufficient photoreceptors to warrant gene therapy”. Hum Gene Ther 2013; 24: 993-1006. – DOI: 1089/hum.2013.153
- Cideciyan A V, Roman A J, Jacobson S G, Yan B, Pascolini M, Charng J, Pajaro S, Nirenberg S, (2016) “Developing an outcome measure with high luminance for optogenetics treatment of severe retinal degenerations and for gene therapy of cone diseases”. Invest Ophthalmol Vis Sci. 57(7):3211-21. DOI: 1167/iovs.16-19586
- Crognale M A, Fry M, Highsmith J, Haegerstrom-Portnoy G, Neitz M, Neitz J, Webster M A, (2004). “Characterization of a novel form of X-linked incomplete achromatopsia”. Neurosci. 21 (3): 197-203. DOI: 10.1017/s0952523804213384
- Deeb S S, (2005). “The molecular basis of variation in human color vision”. Genet. 67 (5): 369-377. DOI: 10.1111/j.1399-0004.2004.00343.x
- Deng WT, Li J, Zhu P, Chiodo V A, Smith W C, Freedman B, Baehr W, Pang J, Hauswirth W W. (2018) ‘Human L- and M-opsins restore M-cone function in a mouse model for human blue cone monochromacy’. Molecular Vision (2018) 24, 17-28. PMID: 29386880.
- Deng WT, Li J, Zhu P, Freedman B, Smith W C, Baehr W, Hauswirth W W, (2019) “Rescue of M-cone Function in Aged Opn1mw−/− Mice, a Model for Late-Stage Blue Cone Monochromacy”. Investigative Ophthalmology & Visual Science August (2019) Vol.60, 3644-3651. PMID: 31469404.
- Buena-Atienza E, Rüther K, Baumann B et al. (2016) “De novo intrachromosomal gene conversion from OPN1MW to OPN1LW in the male germline results in Blue Cone Monochromacy”. Sci Rep 6, 28253 (2016). DOI: 10.1038/srep28253.
- Emily R. Sechrest, Kathryn Chmelik, Wendy D. Tan, Wen-Tao Deng, (2023) “Blue cone monochromacy and gene therapy” Vision Research,Volume 208,2023,108221,ISSN 0042-6989, – DOI: 1016/j.visres.2023.108221
- Gardner J C, Liew G, Quan Y H, Ermetal B, Ueyama H, Davidson A E, Schwarz N, Kanuga N, Chana R, Maher E, Webster A R, Holder G E, Robson A G, Cheetham, M E, Liebelt J, Ruddle J B, Moore A T, Michaelides M, Hardcastle A J, (2014). “A three different cone opsin gene array mutational mechanisms with genotype-phenotype correlation and functional investigation of cone opsin variants”. Mutat. 35 (11): 1354-1362. DOI: 10.1002/humu.22679
- Gardner J C, Webb T R, Kanuga N, Robson A G, Holder G E, Stockman A, Ripamonti C, Ebenezer N D, Ogun O, Devery S, Wright G A, Maher E R, Cheetham M E, Moore A T, Michaelides M, Hardcastle A J, (2010). “X-Linked Cone Dystrophy Caused by Mutation of the Red and Green Cone Opsins”. J. Hum. Genet. 87 (1): 26–39. doi:10.1016/j.ajhg.2010.05.019.
- Gardner J C, Michaelides M, Holder G E, Kanuga N, Webb T R, Mollon J D, Moore A T, Hardcastle A J, (2009). “Blue cone monochromacy: Causative mutations and associated phenotypes”. Molecular Vision. 15: 876–884. PMC2676201. PMID 19421413.
- Haer-Wigman L, den Ouden A, van Genderen MM, Kroes HY, Verheij J, Smailhodzic D, Hoekstra AS, Vijzelaar R, Blom J, Derks R, Tjon-Pon-Fong M, Yntema HG, Nelen MR, Vissers LELM, Lugtenberg D, Neveling K. (2022) “Diagnostic analysis of the highly complex OPN1LW/OPN1MW gene cluster using long-read sequencing and MLPA.” NPJ Genom Med. 2022 Nov 9;7(1):65. doi: 10.1038/s41525-022-00334-9.
- Hanna K, Nieves J, Dowd C, Bender K O, Sharma P, Singh B, Renz M, Ver Hoeve J N, Cepeda D, Gelfman C M, Riley B E, Grishanin R N, (2023) “Preclinical evaluation of ADVM-062, a novel intravitreal gene therapy vector for the treatment of blue cone monochromacy”. Mol Ther. 2023 Jul 5;31(7):2014-2027. doi: 10.1016/j.ymthe.2023.03.011.
- Huddart, J (1777). “An account of persons who could not distinguish colours”. Philos. Trans. R. Soc. 67: 260. doi:1098/rstl.1777.0015. S2CID186212155.
- Katagiri S., et al. , (2018) “Genotype determination of the OPN1LW/OPN1MW genes: Novel disease-causing mechanisms in Japanese patients with blue cone monochromacy”. Sci. Rep. 8, 11507 (2018). DOI: 1038/s41598-018-29891-9
- Keller U, Wissinger B, Tippmann S, Kohl S, Kraus H, Foerster M H, (2004). “Blue cone monochromatism: clinical findings in patients with mutations in the red/green opsin gene cluster”. Graefes Arch. Clin. Exp. Ophthalmol. 242 (9): 729-735. DOI: 1007/s00417-004-0921-z .
- Kohl S, Hamel C P, (2011). “Clinical utility gene card for: blue cone monochromatism”. Eur. J. Hum. Genet. 19 (6): 732. doi:1038/ejhg.2010.232.
- Ladekjaer-Mikkelsen, A S; Rosenberg, T; Jørgensen, A L (1996). “A new mechanism in blue cone monochromatism”. Hum. Genet. 98 (4): 403–408. doi:1007/s004390050229.
- Lewis R A, Holcomb J D, Bromley W C, Wilson M C, Roderick T H, Hejtmancik J F, (1987). “Mapping X-linked ophthalmic diseases: III. Provisional assignment of the locus for blue cone monochromacy to Xq28”. Arch. Ophthalmol. 105 (8): 1055-1059. DOI: 1001/archopht.1987.01060080057028
- Luo X, Cideciyan A V, Iannaccone A, Roman A J, Ditta L C, Jennings B J, Yatsenko S, Sheplock R, Sumaroka A, Swider M, Schwartz S B, Wissinger B, Kohl S, Jacobson S G, (2015). “Blue cone monochromacy: visual function and efficacy outcome measures for clinical trials”. PLOS ONE. 10 (4): e0125700 doi:1371/journal.pone.0125700.
- Mancuso K, Hauswirth W W, Li Q, Connor T B, Kuchenbecker J A, Mauck M C, Neitz J, Neitz M, (2009) “Gene therapy for red-green colour blindness in adult primates”. Nature (2009) 461:784-787. PMID: 19759534.
- Mancuso K, Mauck M C, Kuchenbecker J A, Neitz M, and Neitz J, (2010) “A Multi-Stage Color Model Revisited: Implications for a Gene Therapy Cure for Red-Green Colorblindness’2 (2010) R.E. Anderson et al. (eds.), Retinal Degenerative Diseases, Advances in Experimental Medicine and Biology 664. PMID: 20238067.
- Mascio A A, Roman A J, Cideciyan A V, Sheplock R, Wu V, Garafalo A V, Sumaroka A, Pirkle S, Kohl S, Wissinger B, Jacobson S G and Barbur J L, (2023) “Color vision in blue cone monochromacy: outcome measures for a clinical trial” Transl Vis Sci Techno 2023 Jan 3;12(1):25 DOI: 1167/tvst.12.1.25
- Michaelides M, Johnson S, Simunovic M P, Bradshaw K, Holder G, Mollon J D, Moore A T, Hunt D M, (2005). “Blue cone monochromatism: a phenotype and genotype assessment with evidence of progressive loss of cone function in older individuals”. Eye (Lond). 19 (1): 2–10. doi:1038/sj.eye.6701391. PMID15094734.
- Mizrahi-Meissonnier L, Merin S, Banin E, Sharon D, (2010). “Variable Retinal Phenotypes caused by mutations in the X-linked phopigment gene array”. Inv. Ophthal. Vis. Sci. 51 (8): 3884-92. PMID 20220053.
- Nathans, J (1987). “Molecular biology of visual pigments”. Rev. Neurosci 10: 163-194. PMID 3551758.
- Nathans, J (1999). “The evolution and physiology of human color vision: insights from molecular genetic studies of visual pigments”. 24 (2): 299-312. PMID 10571225
- Nathans, J; Davenport, C M; Maumenee, I H; Lewis, R A; Hejtmancik, J F; Litt, M; Lovrien, E; Weleber, R; Bachynski, B; Zwas, F; Klingaman, R; Fishman, G (1989). “Molecular genetics of human blue cone monochromacy”. Science. 245 (4920): 831–838. doi:1126/science.2788922.
- Nathans J, Maumenee I H, Zrenner E, Sadowski B, Sharpe L T, Lewis R A, Hansen E, Rosenberg T, Schwartz M, Heckenlively J R, Trabulsi E, Klingaman R, Bech-Hansen N T, LaRoche G R, Pagon R A, Murphey W H, Weleber R G, (1993). “Genetic heterogeneity among blue cone monochromats”. J. Hum. Genet. 53 (5): 987–1000. PMC1682301. PMID 8213841.
- Nathans J, Piantanida T P, Eddy R L, Shows T B, Hogness D S, (1986). “Molecular genetics of inherited variation in human color vision”. Science 232 (4747): 203-210. PMID 3485310.
- Nathans J, Thomas D, Hogness D S, (1986). “Molecular genetics of human color vision: the genes encoding blue, green, and red pigments”. Science 232 (4747): 193-202. PMID 2937147
- Neitz J, Neitz M, (2011). “The genetics of normal and defective color vision”. Vision Res. 51 (7): 633–651. doi:1016/j.visres.2010.12.002.
- Neitz M, (2000). “Molecular Genetics of Color Vision and Color Vision Defects”. Archives of Ophthalmology. 118 (5): 691–700. doi:1001/archopht.118.5.691.
- Pinckers A, (1992) “Berson test for blue cone monochromatism”. Int. Ophthalmol. 16 (3):185-186. PMID 1452423.
- Reitner A, Sharpe L T, Zrenner E, (1991). “Is colour vision possible with only rods and Blue sensitive cones?”. Nature. 352 (6338): 798–800. Bibcode:352..798R. doi:10.1038/352798a0. PMID1881435. S2CID 4328439.
- Reyniers E, et al. , (1995) “Gene conversion between red and defective green opsin gene in blue cone monochromacy”. Genomics 29, 323–328 (1995). – PubMed
- Semenov E P, Sheplock R, Roman A J, McGuigan D B, Swider M, Cideciyan A V, Jacobson S G, (2022) “Reading performance in blue cone monochromacy: defining an outcome measure for a clinical trial.” Transl Vis Sci Technol. 2020 Dec 8;9(13):13. – doi: 1167/tvst.9.13.13
- Sloan L L, Newhall S M, (1942). “Comparison of cases of atypical and typical achromatopsia”. American Journal of Ophthalmology. 25 (8): 945–961. doi:1016/S0002-9394(42)90594-4.
- Smallwood P M, Wang Y, Nathans J, (2002) “Role of a locus control region in the mutually exclusive expression of human red and green cone pigment genes”. Proc. Natl. Acad. Sci. U.S.A. 99, 1008–1011 (2002). – PMC – PubMed
- Spivey B E, (1965) “The X-linked recessive inheritance of atypical monochromatism”. Arch. Ophthalmol. 74 (3): 327–333. doi:1001/archopht.1965.00970040329007.
- Sumaroka A, Cideciyan AV, Sheplock R, Wu V, Kohl S, Wissinger B, Jacobson SG, (2020) “Foveal therapy in blue cone monochromacy: predictions of visual potential from artificial intelligence”. Front Neurosci. 2020 Aug 3;14:800. doi: 10.3389/fnins.2020.00800.
- Sumaroka A, Garafalo AV, Cideciyan AV, Charng J, Roman AJ, Choi W, Saxena S, Aksianiuk V, Kohl S, Wissinger B, Jacobson SG. (2018) “Blue cone monochromacy caused by the C203R missense mutation or large deletion mutations”. Invest Ophthalmol Vis Sci. 2018 Dec 3;59(15):5762-72. doi:https://doi.org/10.1167/iovs.18-25280
- Wang C., et al. , Novel OPN1LW/OPN1MW deletion mutations in 2 Japanese families with blue cone monochromacy. Hum. Genome Var. 3, 16011 (2016). – PMC – PubMed
- Wang Y., et al. , A locus control region adjacent to the human red and green visual pigment genes. Neuron 9, 429–440 (1992). – PubMed
- Wang, Y; Macke, J P; Merbs, S L; Zack, D J; Klaunberg, B; Bennett, J; Gearhart, J; Nathans, J (1992). “A locus control region adjacent to the human red and green visual pigment genes”. 9 (3): 429-440. PMID 1524826.
- Weale, RA (1953). “Cone Monochromatism”. The Journal of Physiology. 121 (3): 548–569. doi:1113/jphysiol.1953.sp004964.
- Winderickx J, Sanocki E, Lindsey DT, Teller DY, Motulsky AG, Deeb SS, (1992). “Defective colour vision associated with a missense mutation in the human green visual pigment gene”. Nat. Genet. 1 (4): 251–6. doi:1038/ng0792-251.
- Wissinger B, Baumann B, Buena-Atienza E, Ravesh Z, Cideciyan AV, Stingl K, Audo I, Meunier I, Bocquet B, Traboulsi EI, Hardcastle AJ, Gardner JC, Michaelides M, Branham KE, Rosenberg T, Andreasson S, Dollfus H, Birch D, Vincent AL, Martorell L, Català Mora J, Kellner U, Rüther K, Lorenz B, Preising MN, Manfredini E, Zarate YA, Vijzelaar R, Zrenner E, Jacobson SG, Kohl S, (2022) “The landscape of submicroscopic structural variants at the OPN1LW/OPN1MW gene cluster on Xq28 underlying blue cone monochromacy”. Proc Natl Acad Sci U S A. 2022 Jul 5;119(27):e2115538119. doi: 10.1073/pnas.2115538119.
- Xie B, Nakanishi S, Guo Q, Xia F, Yan G, An J, Li L, Serikawa T, Kuramoto T, Zhang Z. (2010) ”A novel middle-wavelength opsin (M-opsin) null-mutation in the retinal cone dysfunction rat”. Exp. Eye Res. (2010) 91 (1):26-33. PMID: 20371244.
- Yatsenko S. A., et al. , “High-resolution microarray analysis unravels complex Xq28 aberrations in patients and carriers affected by X-linked blue cone monochromacy”. Clin. Genet. 89, 82–87 (2016). – PMC – PubMed
- Zhang Z, Pang J, Xia F, Guo Q, Li L, An J, Zhang L, Hauswirth W W, Yang S, Li Z. “AAV-mediated Gene Therapy Restores Cone Function In A Rat With An M-cone Opsin Deficiency. A Model For Blue Cone Monochromacy”. PMID: 29386880.
- Zhang Y, Deng W T, Du W, Zhu P, Li J, Xu F, Sun J, Gerstner C D, Baehr W, Boye Sanford L, Zhao C, Hauswirth W W, Pang J, (2017) “Gene-based Therapy in a Mouse Model of Blue Cone Monochromacy”. Sci Rep 7, 6690 (2017). https://doi.org/10.1038/s41598-017-06982-7


